自己と非自己を分ける免疫のメカニズム
はじめに
昨今、がん・アレルギー患者や新興感染症の感染者数は増加の一途をたどり、社会的にも免疫学への関心が高まっています。そもそも免疫は、「自分を攻撃せず」、外敵を攻撃するというシステムです。免疫が自己に対して攻撃しないことを免疫寛容といい、免疫寛容はわれわれが生まれた後に一人ひとりが自分でつくり上げています。しかしながら、この免疫寛容がどのように成立しているのか、よく分かっていません。
本研究は、「免疫寛容はどのように出来上がるのか」という問いを発端にすすめられました。

免疫寛容の研究について歴史をひもとくと、1960年のノーベル生理学・医学賞の、ピーター・メダワー博士とマクフラーレン・バーネット博士の「後天的自己免疫寛容」の発見に遡ります。
メダワー博士は、移植を行う際に、生後間もない状態で他人の組織を移植すると生着するのに対し、生後しばらく経ってから移植をした場合、拒絶反応が起こることを示し、この現象を後天的自己免疫寛容と命名しました。
バーネット博士は、一連の免疫応答はリンパ球が中心となって行われており、あらかじめ自己に反応するリンパ球は除去される一方で、「多種多様な病原体を認識する受容体をもつリンパ球が用意されることで、新奇の病原体にでも免疫応答できるシステムが成り立っている」というクローン選択説を提唱しました。
その後、多くの免疫学者により、バーネット博士によるクローン選択説を参考に研究がすすめられ、およそ半世紀をかけ、おおむねバーネット博士のクローン選択説は正しいことが証明されました。
すなわち、我々は生まれた後、リンパ球が個人個人でDNA組換えにより多様な抗原受容体を創り上げ、「自己に反応する成分はあらかじめ除去されることで、それ以外の成分を外敵として反応する」という、獲得免疫システムの基本メカニズムを説明することが可能となりました。
しかしながら、多様な抗原受容体の中から自己に反応する受容体だけをどのように除去するのかについては、いまでもまだよく分かっておらず、現在、免疫学で最もホットな研究テーマの一つとなっています。
_________________________________
脊椎動物は獲得免疫システムと呼ばれる生体防御機構を所持しており、Tリンパ球は獲得免疫システムの中心を担う重要な細胞集団です。すべてのTリンパ球は胸腺と呼ばれるリンパ組織で出来あがり、胸腺内でTリンパ球は、DNA組換えにより10の15乗もの種類の抗原受容体を創り出しています。
しかし、この時に問題となるのが、自己反応性のTリンパ球が創り上げられてしまうことです。この問題を避けるために、胸腺内で自己反応性Tリンパ球は、細胞死が誘導されることで、自己免疫疾患に陥ることを回避しています。
この現象は免疫学の教科書において負の選択と呼ばれています。しかし、胸腺でどのように自己応答性のTリンパ球が除去されるのか、その分子メカニズムは不明な点が多いです。
ことの発端は2001年、胸腺の髄質上皮細胞(medullary thymic epithelial cell; mTEC)は、末梢組織に発現して機能しているはずの遺伝子をタンパク質として発現させていることが示されました(Jenkins et al., Nat. Immunology, 2001)。その後、様々なグループの研究により、われわれのすべての遺伝子は、mTECで自己抗原として発現している可能性が示されました(Samson et al., Genome Res. 2014)。
例えば、インスリンは膵臓で血糖値を下げるために機能していますが、インスリンはmTECでも発現しており、インスリンに対する自己応答性Tリンパ球が胸腺内で除去されることで、1型糖尿病の発症が未然に防がれています。
これまでに、mTECでの自己抗原発現に関わる転写制御因子としてAireが報告されていました(Anderson et al., Science 2002)。実際、Aire遺伝子に変異をもつマウスは自己免疫症状を示します。また、ヒトの場合でも、APECEDと呼ばれる自己免疫疾患になります。しかしながら、AireのみではmTECのすべての自己抗原は制御されていないことが次々と報告されていました。
2015年、今回紹介する論文と同じ東大のグループにより、Aire以外の自己抗原遺伝子を制御する重要なタンパク質として転写因子Fezf2が同定され、AireとFezf2は、それぞれ独自にからだ中の遺伝子を発現させている可能性が示されました(Takaba et al., Cell., 2015)。
しかし、mTECのFezf2とAireはどのように多種多様な遺伝子を自己抗原として発現させているのか、その発現機構の違いは分かっていませんでした。
AireとFezf2はそれぞれ異なる自己抗原遺伝子の発現を制御する
そこでまず、Fezf2とAire間の転写制御プログラムの違いを明らかにするため、野生型マウスとFezf2欠損、またはAire欠損マウスのmTECをフローサイトメトリーにより分取し、RNA-sequencing解析を行いました。
その結果、Fezf2とAireは、それぞれ異なる遺伝子群を発現制御していることが明らかとなりました。また、Aireは主に遺伝子の発現誘導に関わるのに対し、Fezf2は発現誘導のみならず、抑制にも関わっていました。興味深いことに、Fezf2により抑制されている遺伝子の中には、多くのがん抗原が含まれていました。
このRNA-seqデータの結果をもとに、1細胞レベルでのmTECでのAire依存的遺伝子とFezf2依存的遺伝子のmRNAの発現パターンの特徴を解析しました。すると、Aire依存的な遺伝子は、全体のmTEC細胞集団の中の5%未満の細胞集団を単位として遺伝子発現が誘導されており、一定の組み合わせをもちAire依存的遺伝子を発現させていることが解りました(モザイクな発現パターン)。一方で、Fezf2はmTEC全体でFezf2依存的な遺伝子を発現誘導させていることが明らかとなりました(ブロードな発現パターン)。
次に、Fezf2とAireによる遺伝子発現機構を分子レベルで明らかにするために、クロマチン免疫沈降シーケンシング(ChIP-seq)解析とATAC-seq (Assay for transposase-accessible chromatin using sequencing)解析を行った結果、Fezf2依存的遺伝子は転写開始地点(TSS;transcriptional start site)付近がオープンクロマチン状態であり、Aire依存的遺伝子はクローズドクロマチン状態であることが解りました。以上の結果から、Fezf2とAire は転写や翻訳に至る以前のエピジェネティックな段階から、遺伝子の発現制御メカニズムがまったく異なることが明らかとなりました。
Fezf2はChd4と複合体を形成し、自己抗原遺伝子を発現させる
では、Fezf2とAireは、それぞれどのような転写制御機構をもっているのでしょうか?これまでに、Aireは、DNAトポイソメラーゼTopやブロモドメイン含有タンパク質Brd4といったさまざまな転写制御因子と相互作用することで、遺伝子を誘導させていることが明らかとなっていました(Bansal et al., Nat. Immunology, 2017)。
一方で、Fezf2はこれまで相互作用する因子群は報告がありませんでした。そこで、Fezf2タンパク質に対して共役免疫沈降と質量分析解析を行うことで、Fezf2と相互作用するタンパク質をスクリーニングすることを行いました。実際には、ヒト腎癌上皮細胞株の293T細胞とマウス胸腺髄質上皮細胞株の1C6を使用し、それぞれFezf2を過剰発現させ、質量分析により検出されたタンパク質の中で、もっともFezf2との結合に信頼性の高い候補タンパク質として、クロマチン制御因子Chd4(Chromodomain helicase DNA binding protein 4)が挙がってきました。
Chd4はクロマチンの構成やヌクレオソームの位置決定に関わる制御因子として報告されています。Chd4は様々な細胞で、エピジェネティックな発現制御をしていることが明らかとなっていました。免疫沈降法を用いて、mTECのFezf2とChd4は直接的に結合する一方で、AireとChd4は結合しないことが解りました。また、Fezf2-Chd4複合体は、NuRD (Nucleosome remodeling and deacetylase) と相互作用し、Fezf2依存的な遺伝子を発現制御している可能性が示されました。
実際にマウス胸腺において、Chd4がmTECで機能しているかどうかを明らかにするため、胸腺上皮細胞特異的なChd4欠損(cKO; conditional knock-out)マウスを樹立しました。Chd4 cKOマウスではAireとFez2遺伝子の発現が低下していないことが示されました。
次にChd4とFezf2のエピジェネティックな制御機構を明らかにするために、野生型とFezf2 cKO、Chd4 cKOマウスのmTECを用いることでATAC-seq解析を行った結果、Chd4とFezf2は協調的に同じ領域、とくにプロモーター領域のクロマチン状態を制御していることが明らかとなりました。
Fezf2に対するChIP-seq解析の結果からも、Fezf2はプロモーター領域周辺に位置していることが確認されました。以上の結果から、Fezf2はChd4と協調して特定の遺伝子をプロモーター領域周辺で直接的に遺伝子発現を制御していることが明らかとなりました。
Chd4はAire依存的な自己抗原遺伝子の発現にも関わる
以上のATAC-seqとRNA-seq解析の結果から、Fezf2によって制御されるChd4依存的遺伝子は全体の一部であることが判りましたが、残りのChd4依存的遺伝子の発現制御機構は不明でした。しかし、Chd4依存的かつFezf2非依存的な遺伝子リストの中に、Aire依存的な遺伝子が見いだされたことにより、Chd4依存的な遺伝子の発現制御にAireが関与している可能性が示されました。
過去の文献より、mTECのAireはゲノム上のスーパーエンハンサーと呼ばれる制御領域を介してTRAを発現誘導させている可能性が示されていました(Bansal et al., Nat. Immunology, 2017)。そこで、mTECのスーパーエンハンサー領域のATAC-seq解析を行うと、Chd4を欠損するmTECではスーパーエンハンサーのクロマチンアクセシビリティが下がっていました。この結果から、Chd4はスーパーエンハンサーを介して、Aire依存的な遺伝子の発現を制御している可能性が示されました。さらに、1細胞RNA-seqデータを活用し、スーパーエンハンサー近傍遺伝子とAire依存的な遺伝子の共発現パターンを調べた結果、スーパーエンハンサー近傍遺伝子とAire依存的な遺伝子に共発現クラスターが複数同定されました。同様の解析をChd4依存的な遺伝子についても行った結果、こちらでもスーパーエンハンサー近傍遺伝子とChd4依存的な遺伝子を含む共発現クラスターが同定されました。以上の結果をまとめると、Chd4はAireと協調してスーパーエンハンサーを介した遺伝子発現にも関与していることが明らかとなりました。
胸腺上皮細胞のChd4は免疫寛容を誘導する
最後に、Chd4の免疫寛容に関する寄与を調べるため、Chd4 cKOマウスの末梢臓器を調べました。するとChd4 cKOマウスは、Tリンパ球を含んだ炎症性細胞の浸潤が、唾液腺、腎臓、肺、肝臓などの末梢組織で見いだされ、Chd4 cKOマウスの血中より自己抗体産生が検出されました。以上の結果から、胸腺上皮細胞のChd4がTリンパ球の免疫寛容と自己免疫抑制に重要なタンパク質であることが示されました。
まとめ
転写因子Fezf2と相互作用するタンパク質をスクリーニングし、Fezf2と相互作用するクロマチン制御因子Chd4を同定しました。
胸腺髄質上皮細胞のChd4は、転写制御因子AireとFezf2の双方に働きかける重要なタンパク質であり、多種多様な遺伝子を胸腺で異所的に自己抗原として発現させ、自己免疫を抑制していることが明らかとなりました。
以上の結果は、自己と非自己の識別の根源となる免疫寛容の分子基盤の理解を飛躍的に向上させたと考えられます。
リンク
幹細胞のグランドキャニオン?



転写因子のヒストン自体への結合が細胞運命に重要?


バイオステーションはラインボットをリリースします!

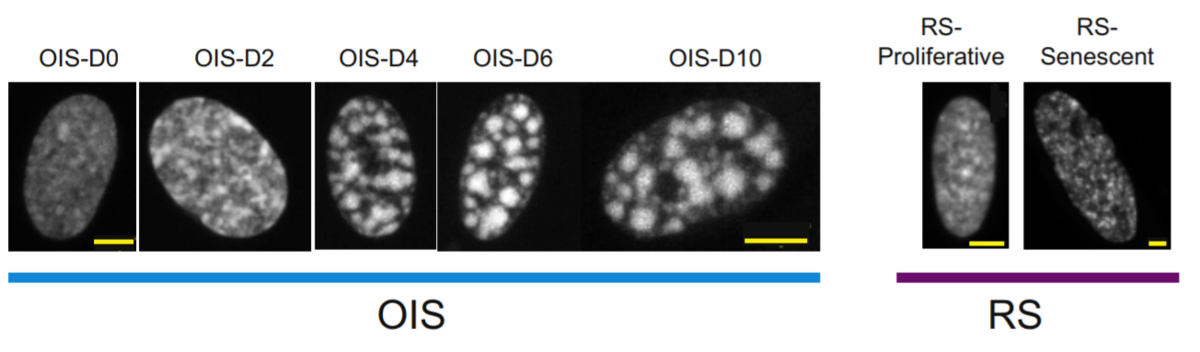
細胞老化に伴う細胞核状態の変化





タンパク質なしにリポソーム内部に特定の物質が濃縮される現象の発見_筆頭著者による論文紹介
今回は筆頭著者の論文紹介として、東京大学大学院総合文化研究科、豊田太郎研究室の杉山博紀(すぎやまひろのり)さんに記事をご寄稿いただきました!ぜひ最後までご覧ください!
----
生命はどのようにして誕生したのか,生命科学に携わる人なら一度は考えたことのある疑問ではないでしょうか.生命の構成単位である細胞の中には様々な生理的な機能が実装されています.無数の生理的な機能一つ一つがあまりに精妙にできていて,いったいどうしたらそれらが一つのシステムとして統合されたものを自発的に組み上げられるのか,これは生物学の最大の謎の一つと言っても過言ではないでしょう.
複数のシステムの統合は差し当たって長い進化の歴史に任せるとして,ある特定の機能一つに限定してみましょう.機能が生まれるには,最低限なにかしらの分子がある程度以上の濃度で区画の内部に存在する必要があります.しかし今日の細胞において,この「濃度を高く保つ機能」はチャネルやポンプなど,複雑な分子システムによって実装されています.そんなシステムがある日偶然現れなくてはいけないのでしょうか.こうしてみると,無数の機能の統合どころか,ある一つの機能が実装された細胞様のシステムを組み上げることさえ困難なように思えます.
今回紹介する論文は,ひょっとするとこのパラドックスに対する一つの回答になっているかもしれません.
具体的には,
細胞サイズのリポソーム*1を自動計測する実験プラットフォームを構築し,実験精度とスループットを両立したデータ取得を可能にしました.さらに本プラットフォームを利用して,酸性リン脂質を含む細胞サイズのリポソームを外部流れ場によって基板表面に押し当てながら特定の分子の水溶液に曝すことで,それらの分子のリポソーム内部の濃度を外水相よりも高く(=濃縮)できること
を発見しました(図1).

この結果は,
- 何もしなければ自発的には濃縮が起こらないリポソームと分子の組み合わせにおいて,外部流れ場というありふれた状況に曝すだけで濃縮を実現できた
という現象としての面白さと,
- 細胞サイズのリポソームを自動で計測できる実験プラットフォームを構築した
という方法論の進展という二つの観点から重要です.
さらに,推測を多大に含みますが冒頭に述べたような,
- 本結果を生命起源や初期進化という観点から眺めると,豊かな視点が広がるかもしれない
という点でもきわめて興味深い現象を発見したのではないかと考えています.
現象自体がめっちゃおもろい
細胞は内部の分子の濃度をコントロールしています.細胞内部のほうが外部よりも濃度が高い状態を維持していることも珍しくありません.これは細胞が,タンパク質などによってATPに蓄えられた化学エネルギーを消費しながら能動輸送をしているからです.しかし,本研究で用いたリポソームには,タンパク質は一切含まれていません.
「濃縮」を非対称な濃度分布の実現と考えれば,たとえば膜を透過できない分子と半透膜を適切に組み合わせることで,ある分子の濃縮が膜を介して進むこともあり得ます.しかし,今回用いた分子の水溶液とリポソームを混ぜて放っておくだけでは,濃縮は起こりませんでした.
しかしこのリポソームを,外部流れ場によって基板と接触させながら特定の分子の水溶液に曝すと,リポソーム内部のその分子の濃度が外部よりも高くなっていったのです.
膜を介した輸送動態が,外部流れ場が引き起こした膜と基板の物理的な接触のみによって質的に変化するというのは驚くべきことです.
さらに,本現象は最初ウラニンという蛍光性の水溶性分子で発見されましたが,蛍光標識されたATPも同様の条件で濃縮できることがわかりました.ATPといえば細胞内のエネルギー通貨としてよく知られたものです.
リポソーム内部に生体機能性分子を内包した,いわゆる「人工細胞」を創成することは構成的アプローチの主要な方法論です.しかしリポソーム膜を介した輸送は通常遅く,人工細胞の機能が長時間持続させられないという点が一つの技術的困難でもありました.ATPが効率的に人工細胞内部に送達・濃縮できることを示した本研究は,長期間に亘って活性を示すような人工細胞の創出につながるかもしれません.脂質膜を介した奇妙の輸送動態という基礎科学的な興味以外にも,人工細胞研究の観点などいろいろな夢が広がる発見といえます.
なんでこんな発見ができたのか?
この現象のポイントは,酸性リン脂質を含むリポソームを用いるという点と,外部流れ場によって基板表面に押し当てながら特定の分子の水溶液に曝すという実験設定です.
しかしある日突然「よし,今日は酸性リン脂質を含むリポソームを作ってそれを基板に押し当てながら適当な分子に曝してみよう!」と思ったわけではありません.思えるわけがありません.
きっかけは,開放骨折でした.
私は修士課程のころはずっと有機合成をしており,博士課程でも合成を主軸にした計画を練っていました.しかし,修論を出し終わった直後に自宅の階段から落ちて,気が付いたら左腕から骨が突き出ていて,運よく1か月くらいで退院はできたのですが(幸い後遺症も残りませんでした),有機合成どころではなくなってしまったのです.何もしないのも暇だったので,計測系でも作っとこうかなと考えました.
近年,微細加工技術を使って観察したい対象に最適化した分析チップを開発する研究が盛んに進められています.これらの分析チップはマイクロ流体デバイスと総称されます.
私はこれまでに,リポソームを捕捉し顕微鏡の一視野内にアレイ化するマイクロ流体デバイスを開発していました(図2).細胞サイズのリポソームの同時並列計測は,従来法ではブラウン運動のせいで技術的に困難でしたが,本デバイスはこれを容易にします.また,リポソームの位置を固定しながら外溶液を交換できます.このため,スループットと精度を両立しながら,高い実験自由度で(どんな溶液をどんな濃度でどんなタイミングで,また,複数種類の溶液をどんな順番で流すか,など)リポソームの刺激応答性を計測できます.

しかし,いろいろなことができるということは,いろいろなことをやらなければならないということでもあります.リポソームのようなメソスケールの分子集合体をきちんと制御する方法論にはまだ暗中模索のようなところも多く,どうしても網羅的な条件探索のようなことが必要になってきます.網羅的な探索といえば聞こえはいいですが,要は何も考えず色々やるということで,あまり人間的な行いではありません.
ということで,マイクロ流体デバイスを使った実験を自動で出来るようにしました(図3).デバイスに溶液を流し入れるためのシリンジポンプ,流す溶液の種類を制御するためのバルブ,観察するための顕微鏡ステージ,光源とシャッターユニット,カメラなどの周辺機器を統合的に制御するプログラムを作りました.このプログラムを,撮影した画像をその場で画像解析するプログラムと組み合わせ,解析結果をもとに系をフィードバック制御出来るようにしました.あらかじめメソッドを組んでおけば,腕を骨折している人でも,たとえば18時くらいにセットアップを終えて(リハビリにおすすめです),Enterボタンを押してプログラムを起動し,家に帰ってご飯を食べて寝て,起きて朝ご飯を食べてラボに戻ってくると,画像解析まで済んだ状態のデータが出ているというようなことができます.

本手法は非平衡状態にある分子集合体の機械支援型同時並列対話型計測(Machine-Assisted Numerous, Simultaneous, and Interactive Observation of Non-equilibrium self-assembly)の略として,MANSIONsと名付けました.広域空間(=大豪邸;mansion)に住んでいるリポソームたちを並列計測することにちなんでいます.
さて,装置のプロトタイプができたら性能試験が必要です.私の開発したマイクロ流体デバイスでは,広域の円形空間にリポソームを一つずつ捕捉します.その後,流れる外溶液を切り替えることでリポソームの外環境を制御する(図2上段参照)のですが,リポソームの捕捉によって外溶液の置換に要する時間が変わるかもしれません.この時間は捕捉されたリポソームの大きさにも依存するかもしれません.そこで,リポソームを捕捉後,外溶液を蛍光性の水溶性分子の水溶液に置換する過程を計測してみたのです.
結果としてリポソームの捕捉の有無は置換に要する時間にはほとんど影響を与えなかったのですが,そんなことよりこの写真を見てください(図4).

明らかにリポソーム内部のほうが背景よりも強い緑色蛍光を示しています.共焦点顕微鏡で見ると,膜(赤)のラインプロファイルは二峰性で中央が暗く,内部の蛍光性分子(シアン)は単峰性で中央が明るくなっています.素直な解釈としては,蛍光性分子は確かに内部に入っていて(膜の外側に堆積しているわけではない),かつ内部の濃度が外部より高いということになりそうです.
しかし実をいうと,初めてこの画像を見たとき私は本当に濃度が高くなっているのだとは思いませんでした.普通に考えればまああり得ないからです.初めてこのデータをボスに見せたときのディスカッションも,こんな見え方になってしまうアーティファクトってどんなものが思いつきますか?というようなものでした.
本現象をそれ以上追及するべきか少し悩みました.いろいろやってみても結局ただの勘違いだったということになるかもしれません.しかし偶然骨が折れていて他にやることがなく,代わりに自動実験装置がありました.どうせ寝ている間にデータを取ってくれるのだし,本当に濃度が高いなんてことがあったらすごいし,装置のパフォーマンステストもかねてやってみるか,みたいなノリで始まったのです.どこにお宝が眠っているのかわかったものではありません.
最近Lab-Automationは様々な文脈で注目を浴びています.その際の論点は,再現性の担保やデータのスケーラビリティ,探索の網羅性などではないかと思います.ただもしかすると,自動化しておくことで妙な現象を本気で検討する際の心理的なハードルが下がるということも,とても大きなポイントなのではないかと思います.
とはいえ自動化されていたとしても,どうやったら本当に濃度が高いと自分を含め相手を納得させられるか,論文を完成させるのには,本当に骨が折れました.
生命起源を考えるとめっちゃめちゃおもろいかもしれない
今日の生命システムが進化的に生じてきたものであるという観点に立って本研究の結果を考えてみると,より豊かな視点が開けるのではないかと私は考えています.
今日私たちが目にする生物はすべて進化によって成立してきたはずです.しかし,生命起源に近い始原的な環境で成立し得るような単純な化学システムに進化可能性を実装するのは,よく考えてみるととても難しい問題です.もしその実装にそんなに選択肢がないとすると,生命起源における細胞様の化学システムの設計原理は,今日の細胞にまで通底する最も基本的なシステムの設計原理を捉えているかもしれません*2.
興味深いことに,今回の濃縮には分子依存性があります.
酸性リン脂質というのは,言い換えればアニオン性のリン脂質ですが,今回の研究の範囲で濃縮が確認された分子はいずれもアニオン性の電荷をもつ分子でした.他方,アニオン性でない分子の水溶液を用いた場合や,アニオン性のリン脂質を含まずに形成させたリポソームでは濃縮は確認されませんでした.
上述した通り,最初期の生命様の化学システムに,どのようにして進化可能性を実装し得たのかというのは大きな謎です.進化可能性の実装には少なくとも何らかの機能が必要であり,機能が生まれるためにはある程度の分子濃度,すなわち濃縮された状態が必要です.
もしも濃縮にはある特定の電荷が必要だ(あるいは膜の電荷との組み合わせ)というようなことになったらどうでしょうか.生命のような複雑なシステムを最初に作るときには,電荷のそろった膜と基質の組み合わせでしか第一歩を踏み出せないとすれば,後々のシステムの設計思想にも大きな影響を与えそうです.
そう思ってみてみると,生体内の重要な機能性物質,たとえばATPとかDNAとかってアニオン性だよな...そういえばアミノ酸の等電点って7以下のものが多いような...
本当でしょうか.
本当だったら面白いですね.
---
(補足1)リポソーム
リン脂質が水中で袋状の二分子膜を形成した分子集合体.イメージとしては,細胞から細胞質,オルガネラ,その他諸々を除去して,最外層の膜構造だけにしたもの(下図).

ドラッグデリバリーの担体としての応用的利用のほか,構成生物学(補足2も参照)の文脈では,細胞の化学モデルの足掛かりとしてよく利用される.特に本研究では,原始細胞のモデルと見立てている.
(補足2)構成的なアプローチと生命起源と進化と濃縮
(1)構成的アプローチ
生物学の伝統的な方法論は,複雑な系を要素に分解して考える還元的なアプローチといえます.一方,この20年で,細胞の「設計原理」の理解を目指す動きが活発化してきました.本研究は,細胞様の化学システムを構成し,単純化されたモデル系を実験的に調べるという,構成的アプローチの中に位置づけられます.
構成的アプローチの問題意識を説明する際,しばしば,コンピューターを理解するにはどうするか,というアナロジーで語られます.チップセットを調べたり,とりあえず分解してみて使われているねじの品番を調べたりという行為が生物学の伝統的な還元的なアプローチに対応します.
しかし,この例でいえば,チューリングマシンを理解することなしに「コンピューターを理解した」と思う人は,まずいないでしょう.とすると問題は,チップセットを調べるだけで,果たしてチューリングマシンにたどり着けるだろうか,ということです.
分解して調べるだけではなく,今あるコンピューターのようにふるまう系を構成してみようとすることで,初めてわかることもあるのではないかというのが根底にある一つの問題意識といえます.もちろん構成的アプローチは,分解して理解するというフェーズをすでにある程度経ているからこそできることであり,いわば還元的なアプローチと相補的な方法論ととらえることができます.
(2)構成的アプローチと進化
作ってみると簡単に言っても,現在の細胞は素晴らしく精妙にできているので,すべてを模倣することは(少なくともしばらくは)不可能です.何に着目してどう実装するか,何をもってモデルの良し悪しを判断するのか.明快な道筋はありません.
一つのありうる方向性として,シンプルな系に進化可能性を実装するにはどうしたらよいか?という問いがあると私は考えています.今日の生命システムたちはある日突然生じてきたのではなく,連綿と続く長い進化の歴史の中で徐々に形成されてきたものです.逆に言えば,今日の生命システムに至るすべての過程において,「進化可能であるべし」という制限が要請されていたはずです.どんなに素晴らしい機能を備えていても,進化可能性を失ってしまったらその時点でそのシステムの歩みは止まってしまうからです.この進化可能性への要請は原始地球に初めて生じた最初期の生命様のシステムにとっても同様のはずです.しかし,進化可能な最初期の生命様のシステムとして,どんな実装がありうるでしょうか.もしその実装にそんなに選択肢がないとすると,それ以降のあらゆるシステムはこのシステムを進化的に拡張しながら成立していくわけですから,始原環境での設計原理は,もしかすると今日の細胞にまで通底する最も基本的な設計原理になっているかもしれません.
こうした問いにも,やはり作ってみながら考えるのがよさそうです.
(3)生命起源と濃縮
濃縮と生命起源の関係についても考えてみます.どんなシンプルな系を想定するにせよ,進化可能性が宿るということは何らかの機能があるということです.機能が宿るには,それなりの濃度の分子が,細胞のような区画に閉じ込められている必要があります.外水相の濃度が高ければ,受動輸送で区画の中にもそれなりの濃度の分子が入るでしょう.でも,そんな状況だったら区画はもはや必要ないし,外部にそれだけの分子が既に存在しているという状況も不自然です.能動輸送を実現する膜タンパク系が存在していれば問題は解決します.タンパク質だけなら,偶然に生成されてもいいかもしれません.しかし,タンパク質がきちんと働くには,それと協調した膜システムが不可欠です.そんな複数の要素が互いに依存しあうようなシステムはどうしたら生じるのでしょう.シンプルなシステムから進化できればいいのですが,今問題にしていたのは,まさにどうしたら進化できるかというところから話が始まっており,トートロジーが発生しています.
本研究で示したような,非生物的な過程のみで細胞様の区画の中に濃縮できるということは,このトートロジーのスタート地点を覆せるかもしれません.
-----
バイオステーションでは、引き続き筆頭著者による論文紹介を募集しています!
詳細はこちらをご覧ください(リンク)。ご連絡お待ちしております。
ヒストン修飾と相互作用する新規タンパク質の網羅的同定



