ウィーン滞在記(1)
今回は論文紹介ではなく管理人の雑記になります。
管理人が2021年4月から3か月ほどオーストリアのウィーンに短期滞在しておりますので、その様子などをお伝えすることで皆様のお役に立てればと思います。
今回は渡航までの経緯などをご紹介しようと思います。
---
なんとなくきっかけからですが、管理人は博士課程も終盤がみえてきて、そろそろ次のキャリアを考えなければならないという気持ちがありました。
海外でポスドクがなんとなくいいのかなぁと思っていたのですが、海外のPIにコンタクトとるにも海外学会もオンラインになってしまったしどうしよう、、となかなか困った状況でした。
そこでラボの皆さんの紹介などもあり、学術振興会の海外若手挑戦プログラムというので数か月海外で研究してみよう、という運びになりました(昨年の夏ごろでした)。
この海外若手挑戦プログラムというのが、
・採用時に博士課程に在学していれば応募できる(DCを持っていなくてもOK)
・金銭的にサポートがしっかりしている(100~140万円+渡航費)
・比較的競争が緩い(自分の年度/分野では採択率50%)
というので結構よいように思います。
公式のページのリンクは以下です。
----
このプログラムにだそうと決めたのはいいのですが、意中の研究室が1つに決まっていたわけではないので、ここから受け入れて頂く研究室を選び始めました。
研究室選びでは、はじめにこれまでにいいな、と思っていたラボを中心にラボの様子を調べ、4研究室に絞りました。
次にこの中から、分野が近いラボは視野が広がりづらいというアドバイスに基づき分野がある程度離れる2研究室に絞り、諸条件を加味して1研究室に連絡を取ってみることにしました。
結果、奇跡的にOKの返事を頂くことができ、あとは申請書が採択されれば受け入れていただけるということになりました。
---
というわけで、渡航前年の8月末に申請書を作成しました。(年度ごとに8月〆切と3月〆切の2回の応募があります。)
詳しくは学術振興会のページを参考にしていただくのがよいですが、今年度はこれまでの研究1ページ、渡航先でやりたいこと1ページの内容でした。
(DCやPD、海外学振と比べたら全然軽いですね、、まあ渡航期間も短いですし審査も大変でしょうからこれくらいがいいのかもしれません)
ただ私は受け入れ許可のメールをいただいた時点で申請書締め切りまで1週間を切っていたので渡航先でやりたいことを書くのは苦労しました。
結果的にはメールだけのやり取りで細部を詰め切ることができなかったのでよくなかったですね、、Zoomで話したのは申請書を書いた後でした、、
渡航先でやりたいことは先方とよく話し合って書けるように前々から準備されるとよいと思います、、
---
そんなわけでなんとか申請書を提出しまして、12月末に採択内定通知を頂きました。今年はコロナの影響か応募人数が少なかったのも幸いしたように思います。
このあと1月以降で先方のメンターさんやボスと数回Zoomでディスカッションをして具体的にどんな実験をするかについて詰めました。
もちろんラボによると思いますが、私の受け入れ先は何やってもいいからアイデアだしてみて!という感じだったので結構頭を悩ませました。
最終的には先方の提案から自分がいくつかピックアップするような形でやることを決めていきました。
また、この採択決定後から渡航までは、受入れラボのテーマに近い論文をウォッチすることで少しでもその分野の知識を入れておこうと試みました(もっと気合い入れればよかった、)。
---
プログラムをスタートする時期はある幅の中で自由に決められるのですが、私は受入れラボとの話し合いの結果4月に渡航することになりました。
そういうわけで4月に、飛行機も何度か変更されたりしてやはり平時ではないなあというのを感じつつも、なんとかウィーンに渡航することができました。
渡航後も5日間隔離されたのちにテストを受けて陰性を確認してからラボへの訪問を始めました。
研究生活については今回の記事である程度反響があればウィーン滞在記(2)として書いてみようかと思います。
---
ちなみに町はいかにもヨーロッパでおしゃれな雰囲気で、なんだか観光客のような気分になりました。(残念ながらコロナのせいで観光地どころかレストランもやっていませんが、)
一応2枚ほど写真でご紹介します
・シュテファン聖堂というウィーンのシンボル的建物

・中心地近くの街並み

ひとまず以上になりますが、若い読者の皆様にこのようなプログラムがあることを知っていただければ嬉しいです。(ご質問などありましたら何かでご連絡ください。)
では。
DNAの傷が転写を活性化する?
細胞の中でDNAはヒストンやその他たくさんの非ヒストン性クロマチン因子によってパッキングされている。
これらの因子によるゲノム構造の制御が、正常な遺伝子発現のコントロールには重要である。
この中でHMGA2という因子が胎生期の細胞やガン細胞などで高く発現し、結構重要らしいことが長年の研究で分かってきている。
しかしながら、HMGA2の働く分子メカニズムは(その重要性の割に)あまり統一的な見解が得られず、グループ毎に各々が言いたいことを主張しているような感じである。
筆者らのグループはこれまでにHMGA2がDNA損傷応答関連因子と相互作用することを報告していたが、具体的に転写制御まで行きつく分子メカニズムはよく分かっていなかった。
今回はなんとHMGA2がDNAに傷を入れることでDNA損傷応答関連因子を呼び込み、転写を活性化するというモデルを提唱する報告を紹介する。

----
そこではじめにHmga2をノックアウトした条件でγH2A.XのChIP-seqを行った。
(ほんとにγH2A.XのChIP-seqってこんなにTSSに濃縮するのかな、、??ご存知の方いらっしゃったら教えて頂きたいです、、、)

というわけで、Hmga2はγH2A.Xの呼び込みに重要である可能性が示唆された。
では、Hmga2は転写にどのような影響を与えるのだろうか?
筆者らはさらにHmga2のノックアウトマウスにおいて転写の開始を見るリン酸化ポリメラーゼIIのChIP-seqを行った。
この結果、Hmga2のノックアウトによってリン酸化ポリメラーゼII(pPol2)のTSSへの濃縮も減少することが分かった。
(ちなみに遺伝子発現も一部の遺伝子群で減少することを見ている)

というわけで、どうやらHmga2は(γH2A.Xの呼び込みを介して?)転写を正に制御するように見える。
----
では、Hmga2はどのようにγH2A.Xの呼び込みを制御しているのだろうか?
筆者らは細胞をMNaseで処理してスクロース勾配法で分画することでHmga2と同じ画分に存在するタンパク質を網羅的に解析した(若干この流れはストレートでない感じがしたが、)。
この結果ヒストンシャペロンであるFACTがHmga2と同じ画分に存在し、Hmga2のノックアウトでその分画から減少することが分かった。
(このデータからはすべて標的遺伝子を3つに絞った解析)
---
以下細かいので端折ってしまうと、(ごめんなさい)
Hmga2がDNAに一本鎖切断を入れる(これは以前報告がある)→FACTがやってきてγH2A.Xを呼び込む→DNA損傷応答依存的DNA脱メチル化→転写活性化というモデルらしい。

----
管理人コメント
・ぶっちゃけなんか不安なデータが多い。MNase-seq、γH2A.XやHmga2やFACTのChIP-seqってこんなピークの出方で正しいのかな、、、しかも後半は見る遺伝子3つに絞っているし、どれだけ一般的なメカニズムなのかはよく分からない。
・ただクロマチン因子がDNA損傷を入れて転写を活性化する、というのはコンセプトとしては面白いかも?
Positioning of nucleosomes containing γ-H2AX precedes active DNA demethylation and transcription initiation
Stephanie Dobersch, Karla Rubio, Indrabahadur Singh, ..., Dulce Papy-Garcia & Guillermo Barreto
脊髄の再生を促進するグリア細胞の性質変化
今回紹介する論文では、ゼブラフィッシュの系を用いてグリア細胞の上皮間葉転換(Epithelial-to-Mesenchymal Transition, EMT)が脊髄損傷後の再生に重要であることを見出した。
我々哺乳類とは異なりゼブラフィッシュは高い再生能を有し、脊髄が損傷しても6-8週で元通りにすることができる。
これまでに今回の筆者らのグループなどは、ゼブラフィッシュの脊髄再生には特殊なグリア細胞集団が損傷部位を架橋することが重要であることを報告してきた。

(哺乳類では中枢神経においてはアストロサイトが損傷に反応し再生ではなく傷口をふさぐような機能を果たすため再生ができないと考えられている。また、哺乳類の末梢神経系ではシュワン細胞が損傷部位を架橋することで再生を促進することが知られている。)

----
筆者らはこれまでに損傷部位の架橋を行う特殊なグリア細胞集団に発現するマーカー遺伝子としてctgfaを見出してきた。
そこではじめに損傷時においてctgfa陽性細胞集団を分取しRNAseqを行うことで、この細胞集団がどのような遺伝子発現プロファイルをしているのかを網羅的に解析した。
すると興味深いことにこのctgfa陽性細胞集団では上皮系の遺伝子発現が減少し、間葉系の遺伝子発現が上昇していることが分かった。
このような遺伝子発現変化は上皮間葉転換と言われ、"上皮細胞がその細胞極性や周囲細胞との細胞接着機能を失い、遊走、浸潤能を得ることで間葉系様の細胞へと変化するプロセス(Wikipedia)"として有名である。
さらに組織染色からもEカドヘリンの減少とNカドヘリンの増加という上皮間葉転換にみられる代表的な変化が起きていることからも、グリア細胞集団による損傷部位の架橋では上皮間葉転換様の変化が起きていることが明らかになった。
----
そこで気になるのが、この上皮間葉転換様の運命転換はグリア細胞による架橋、そして脊髄再生に重要なのか、という点である。
この問いに迫るため筆者らはCRISPR/cas9システムを用いたノックアウトスクリーニングを行った。
このスクリーニングでは受精卵においてCRISPR/cas9を用いて遺伝子をノックアウトし、大人になって損傷を加えることでグリア細胞による架橋と脊髄再生による遊泳能力の改善を評価した。
その結果、上皮間葉転換を制御するいくつかの遺伝子の欠損でグリア細胞による架橋と脊髄再生による遊泳能力の改善が弱くなることを見出した。
----
ただ再生できなくなる変異体なら取れなくもなさそうだが、上皮間葉転換を促進するだけで再生も促進することができるのだろうか。
このために、Twistという上皮間葉転換を促進する遺伝子を再生時に発現させるゼブラフィッシュを作成し、再生能を評価した。
すると興味深いことに、Twistを再生時に強制発現させると、グリア細胞による架橋が促進され、再生能も向上することが明らかになった。
ということで、上皮間葉転換を促進するだけでも脊髄再生が促進される可能性が示唆された。
---
まとめると、今回の論文でゼブラフィッシュの脊髄再生ではグリア細胞が上皮間葉転換様の性質変化を起こし、損傷部位を架橋することが正常な再生に重要であることが分かった。
哺乳類では脊髄に同様の機能を持つ細胞はいないため、このような細胞集団を生みだすことが脊髄の修復には重要なのかもしれない。

----
コメント
・上皮間葉転換と言えばガンとかで有名な気がするが再生にも関わっているのか。哺乳類で再生能が落ちているのはガン化のリスク下げるためなのかなぁ。
・上流がYAPらしいが再生時に物理的な圧力を感知したりできるのか?物理ストレスを感知して再生するとかなら結構面白い。
・応用的には哺乳類で再生ができると嬉しい。部分的に上皮間葉転換をおこせれば、って感じかな。
今回の論文
Localized EMT reprograms glial progenitors to promote spinal cord repair, 2021
ゲノム中の塩基配列の偏りの意義?
生物のゲノムはATGCの4つの塩基の組み合わせからなる。
このA, T, G, Cはゲノム中に均等に存在するわけではなく、ある一定の偏りを持って並んでいる。
では、この塩基の配列の分布の偏りは何か役割を持つのだろうか?
これまでCG配列に結合する因子とその機能がいくつか報告されてきた。
このためCG配列が多いことには何らかの意味があることが示唆されてきたが、AT配列については比較的その意義は調べられてこなかった。
今回、ゲノム中のATが濃縮する配列を読み取り、細胞運命を制御する因子を同定した論文を紹介する。
筆者らはまず、ATが多いDNAに結合する因子を網羅的に探索した。
この結果、これまでATに結合することが知られていた因子を含め、複数の因子がヒットした。
筆者らはこの中からSall4という因子に着目した。
SALL4は神経/四肢などの発生に重要な因子で、ヒトではオキヒロ症候群という疾患と関連していることが知られている。
----
ではSall4がATに多い遺伝子座に結合することは遺伝子発現にどのような影響を与えるのだろうか?
この結果興味深いことに、Sall4の機能欠損ではATが多い遺伝子の発現が上昇し、Sall4の過剰発現ではATが多い遺伝子の発現が減少することが分かった。
このことからSall4はATが多い遺伝子の発現を抑制している可能性が示唆された。
----
さらに筆者らはSall4がATの多い遺伝子に結合する生物学的意義を探索した。
このために、筆者らはSall4のDNA結合部位に点変異を導入したマウスを作成した。
Sall4のノックアウトマウスは胎生致死になることが知られていたが、なんと今回作成された点変異マウスも胎生致死になることが明らかになった。
このことから、Sall4がATの多いゲノム領域に結合することは正常な初期発生に重要である可能性が示唆された。
----
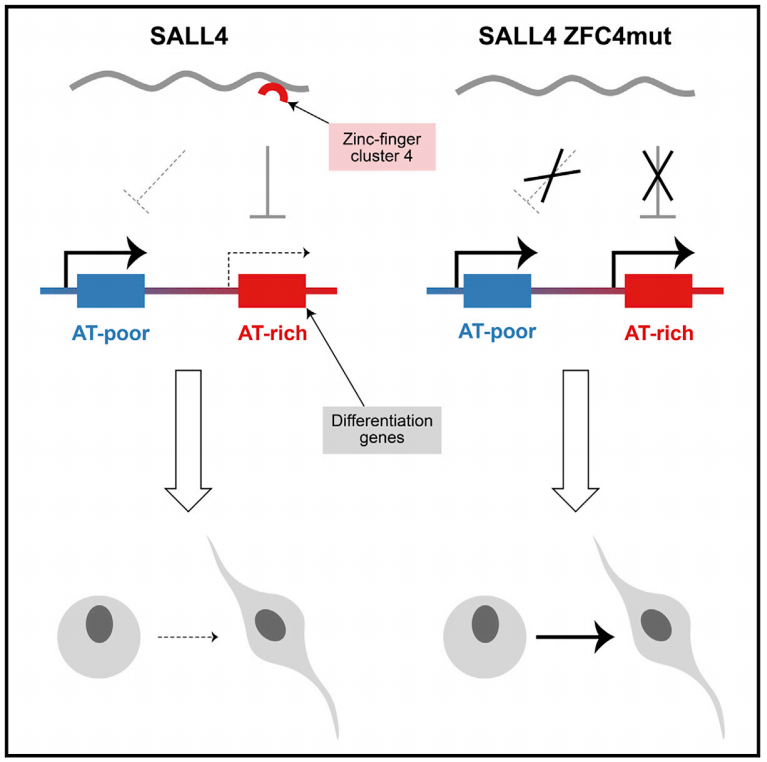
結果は以上のような感じで、今回の結果から
・Sall4がATに偏ったゲノム領域に結合し、遺伝子発現を抑制することで正常な発生を可能にする
というモデルを提唱している。
彼らのメッセージとしては
・それぞれの遺伝子座に固有のゲノム配列から遺伝子を個々に制御するより、塩基の分布の偏りを読み取ってまとめて遺伝子発現を制御するほうが合理的だよね
ということらしい。まあそうかもしれない。
以下グラフィカル
----
----
コメント
・Sall4の(AT結合ドメインの)機能解析を"ゲノム中のATの偏りの意義"と概念的に発展させているのは勉強になった。やはり一般的なメッセージにするのは大事だな。(結果から十分サポートされているかはやや疑問)
イマイチわからなかったこと(解決した方は教えてください!!)
・Sall4結合領域と発現制御する遺伝子の遺伝子座の関係が不明瞭。載っているデータでSall4結合領域→AT rich、Sall4変異で発現上昇→AT richはいいが、Sall4変異で発現変動⇔Sall4結合は不明。いかにもSall4が直接AT rich geneを抑制しているように書いているが、間接的な可能性もあるのでは?
・ATの比を反映して遺伝子発現制御と主張しているが、Sall4がATの比だけでゲノムにターゲティングしているとは思えない。ATはクロマチン閉じている印象だが、彼らはSall4はオープンクロマチンに濃縮することも示しているしSall4の結合には配列以外の因子も関係ありそう。"ATの比を反映して"というにはデータ薄い?
SALL4 controls cell fate in response to DNA base composition, Mol. Cell, 2020
2020年生命科学研究まとめ&2021年展望
学生の漫談みたいな感じなのであしからず。昨年の2019年まとめ&2020年展望はこちら。
全体のまとめ、神経幹細胞研究、クロマチン研究という感じの流れです。最後にバイオステーションの来年の目標とお願いがあります。
全体
情勢を反映して新型コロナウイルス関連の論文はめちゃくちゃ多かったですね。本当に文字通り毎週三大誌に載っていた気がします。正直全然内容はフォローできていないのですが、喫緊の事態になると爆発的に研究が進むんだなと思いました。日本からの論文は少なかったように感じるのは残念です。しばらくは新型コロナウイルス関連論文はこの勢いで報告されていくのではないかと思います。
また新型コロナウイルスの影響でオンラインセミナーが多かったのは良かったです。神経系とかクロマチン関連とか各分野オンラインセミナーをやっていました。普通なら国際学会でも行かないと聞けないような話をたくさん聞けるのは良かったですね。こういった試みはコロナが収まっても続いてほしいなぁ。
流行っていた研究領域は、うーん、よく分からないですね。クライオEMによる構造解析はトップジャーナルにすごくたくさん出ていましたが、管理人があんまりすごさを理解できていないのでコメントしづらい、、というわけで、管理人が比較的理解できる分野のまとめに移ります。
神経幹細胞研究(特に胎生期)
今年はトップジャーナルに神経幹細胞研究は全然載りませんでしたね、、2019年はそこそこ出ていただけにどうしたんだという感じですが、、胎生期神経幹細胞研究で三大誌に載ったのはSong Hai ShiのNatureとPierre VanderhaeghenのScience、Daniel A. LimのScienceだけかな(ぎりぎりARHGAP11Bとバイステでも紹介したPurterb-seqも?他にあったら教えて下さい!)。どれも最先端のスーパーテクノロジーというわけではない古典的な実験で新しい概念を提唱しているのはすごいと思います。
全体のトレンドとしては純粋な神経発生だけではちょっと苦しくて、進化とか疾患とかと結びつけていかないとなあ、という感じでしょうか。もちろんまだまだ分かっていない重要な課題はたくさんあるので来年以降も神経幹細胞関連の面白い論文に期待です。
日本からは、松崎研の論文が年始に発表されていました。神経幹細胞の分裂様式と神経幹細胞の足(Apical endfoot)の動きの関係をライブイメージングやFRETなど(少なくともこの分野では簡単にできる実験ではない)綿密な実験で示していてとても自分にはできないすごい研究だと思いました。みなさま、大変おめでとうございます。
あとやはり後藤研からの大脳の神経幹細胞の背腹軸決定にエピジェネ因子が関わるという報告でしょうか。背腹軸制御はとっても大事なだけにこれまでShh、WntやBMPなどスター因子の関与はたくさん報告されてきたものの、それらがどのように制御されているのかは不明でした。今回はヒストン修飾に関与するポリコーム因子群がWnt、BMPの上流となり背腹軸決定に貢献することが示されました。背腹軸の異常という想定していなかった表現型を見出すところから始まり、メカニズムまで緻密に詰められた超絶スーパーなお仕事です。
(他にも宮田研のLzts1とか難波さんのARHGAP11Bとかもあったなーと思ってたらどちらも2019年でした、、、記事をご覧の方で自分も胎生期神経幹細胞で2020年に論文出したぞ、という方はご連絡いただけると幸いです。)
クロマチン関連
クロマチン関連といっても分野広すぎですよね、、、管理人がフォローできる範囲だと、今年は"ヌクレオソーム+何かの因子"の構造解析がたくさんトップジャーナルに掲載されていました。SOX2, OCT3/4, cGAS(5報同時)、PARP、NSD、DNMT、BAF(2報)とかですかね(もっとあった気もする)。今後は"ヌクレオソーム+何か"に留まらない構造解析とかがメインになってくるのでしょうか。
個人的興味もあるかもしれませんが、タンパク質分解システムを用いてクロマチン因子の急性の欠損がゲノム構造に与える影響を解析する論文も多かったように思います。昔紹介したNELFとか、プレプリにHP1とかも上がっていた気がします(他にもポリコームは前からやられている気がする)。ここ何年かで代表的な因子がやられつくしていくのではないかと期待しています。
また良くも悪くも印象に残ったのはRichard YoungのMeCP2がphase separationするやつでしょうか。直前にCell Researchに同じような論文が出ているのにNatureに通してしまうのはさすがという感じです。しかもMeCP2がphase separationしますという結果と疾患の点変異だけでよくNatureだなと。まだ魔法の言葉phase separationの効力は少し残っているようです。
バイオステーション来年の目標とお願い
今年もご愛顧いただきありがとうございます。バイステ、来年の目標が一つとお願いが二つあります。
目標:もう少し紹介できる論文の幅を広げる!
宣言しとけば頑張れるような気がしなくもないので宣言しました。頑張ります。
一つ目のお願い:バイステよく間違えたこと書いているので気づいた方はお伝えいただけると幸いです!
(もちろん間違いはなくしたいですが、、)できれば優しめの表現でお願いしたいです笑。
二つ目のお願い:なんか色々バイステ手伝ってくださる方いたら教えてください!
自分も論文紹介してみたいとか、バイステのブログのフォーマットダサいから作りたいとか、バイステの間違いがひどいから添削したいとか、そういう感じのことで何かありましたらご連絡ください。直接言ってもらっても、メールでもツイッターのDMでもなんでもいいです。
というわけで、来年もよろしくお願いします!!
自閉症リスク因子が脳発生に与える影響を一網打尽に!
私達の遺伝情報をコードするゲノムの変異は疾患に結び付くことが知られている。
しかし、ノックアウトマウスの作成および解析は大変な労力が必要であり、多数の細胞種からなるヒトやマウスの体において、遺伝子の欠損がどの細胞に影響を与えるのかを調べるのは未だ簡単ではない。
そこで今回、シングルセルRNAseqとCRISPRを組み合わせた手法により、複数の自閉症リスク因子遺伝子が脳発生に与える影響を細胞種ごとに解析した論文を紹介する。
-----

自閉症は、対人関係の特異性やコミュニケーションの質的な障害などが見られる疾患(?)であり、これまでヒトを用いた研究により多数のリスク遺伝子が同定されている。
これらの遺伝子が、異なる特性を持つニューロンや、アストロサイト、オリゴデンドロサイトといった多数の細胞種からなる脳のどの細胞に影響を与えるのかを網羅的に調べるのは難しかった。
そこで筆者らはシングルセルRNAseqとCRISPRを組み合わせたPerturb-Seqを用いることで複数の遺伝子が神経発生に与える影響を解析することを試みた。

実験の流れは上の図のような感じ
ざっくりとは、
↓
Cas9を発現するマウスの胎児の脳にインジェクション(ウイルスが導入された細胞では標的遺伝子がノックアウトされることが期待される)
↓
ウイルスが導入された細胞をソートし、シングルセルRNAseq
↓
ウイルスにつけていたバーコードの情報から、それぞれの遺伝子を標的としたウイルスが導入された細胞を同定
↓
ノックアウトによって変動した遺伝子発現プロファイルの解析
という流れ。
(ちなみにメインFigureのデータだけで18セットのシングルセルRNAseqしているようだ。お金持ち!)
そういうわけで、35種類の遺伝子のノックアウトウイルスを導入した際にどの細胞種の遺伝子発現プロファイルが変動するかを検討した。
 興味深いのが赤丸で囲ったAnk2, Chd8, Gatad2bの3遺伝子である。
興味深いのが赤丸で囲ったAnk2, Chd8, Gatad2bの3遺伝子である。
これらの遺伝子は、これまで影響があるとは思われていなかったAnk2は抑制性ニューロン、Chd8とGatad2bはオリゴデンドロサイトの遺伝子発現プロファイルを変化させていることが分かる。
実際筆者らは、Chd8の(普通の)ノックアウトによってミエリンの形成が異常になる可能性を見出している。
(これはバイアスなしに変化を記述するPerturb-seqの強みがよく出ている結果のように思える)
最後はヒトの患者でみられた遺伝子発現変化が今回の系でも一部見られることを出している。(まあこれは解析の方法次第でどうにでも見せられそうだが)
------
コメント
・誰にでもできる、というほど簡単な手法ではなさそうだが、今後こういった網羅解析はより一般的になってくるのだろう。細胞種も比較的多い脳で、これまたリスク遺伝子も多い自閉症をモデルにこんなこともできる、というのを示した点では重要だと思った。
・こういう網羅解析は何段階ものクオリティコントロール、正規化、統計処理を重ねるため、こねくり回せば"ポジっぽい"Figureにできてしまうのではないかという懸念はある。やはり最終的には普通のノックアウトマウスなどの機能解析が必要だろう。
・今回は変化が見られる可能性が大きそうな候補因子だけに絞っていたが、もっと多数の遺伝子を対象とすることでこれまで脳発生に関係するとは思われていなかった遺伝子の寄与などを調べられると面白いかもしれない。
In vivo Perturb-Seq reveals neuronal and glial abnormalities associated with autism risk genes, Science, 2020
Xin Jin, Sean K. Simmons, Amy Guo, ..., Aviv Regev, Feng Zhang, Paola Arlotta
相分離を駆動する"ヌクレオソームコア"の構造変化
クロマチンは一般に緩い構造を取ると遺伝子発現が活性化しやすく、凝集した構造を取ると遺伝子発現が抑制されやすい。
このようにクロマチンの凝集度の制御は、遺伝子発現の制御にとても重要である。
これまで、クロマチンの凝集を制御する代表的な因子としてHP1(Heterochromatin protein 1)という因子が知られてきた。
しかしながら、HP1がどのような分子メカニズムで相分離を引き起こしているのかは不明である。

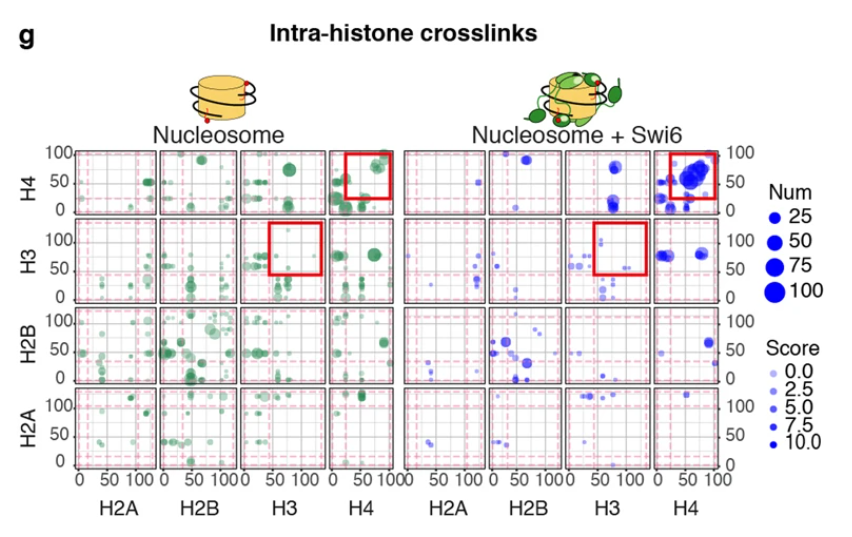
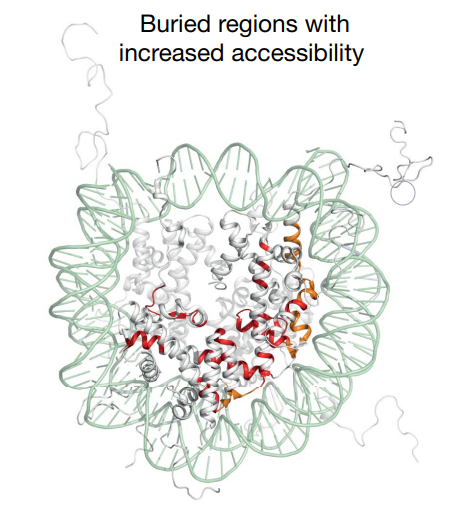
このとき興味深いことに、HP1の存在下ではH3とH3、H4とH4のように、コアヒストンの相互作用にも変化が起きていることが分かった。
これは、HP1によってコアヌクレオソームの構造が変化している可能性を示唆する。
---
---
では、このヌクレオソームの構造変化はHP1の機能に重要なのだろうか?
また構造変化が起きにくいヌクレオソームを用いた場合、HP1による相分離能も低下していることが示された。
---
-----
コメント
・たしかにヌクレオソームの構造自体で凝集状態や相分離の状態が変わるというのは概念的に新しいかも。因果関係はヒストン変異体の実験しかないので、"ヌクレオソームの構造変化→凝集/相分離"がどれだけそれらしいかは疑問が残るが。
・細胞内でも同じようなことが起きているのか?というのはとても気になる。今回の場合、"ヌクレオソームの構造変化は起こさないHP1の変異体"が取れるとよいのだが、難しそう。
---
今回の論文
HP1 reshapes nucleosome core to promote phase separation of heterochromatin, Nature, 2019
S. Sanulli, M. J. Trnka, V. Dharmarajan, R. W. Tibble, B. D. Pascal, A. L. Burlingame, P. R. Griffin, J. D. Gross & G. J. Narlikar