ATAC-seqの歴史
遺伝子の発現がどのように制御されているか知ることは、生物学の根幹である。
これまでの古典的な研究で、遺伝子の発現はプロモーターやエンハンサーの活性で制御されることが分かってきた。
さらに最近、このプロモーターやエンハンサーのクロマチン(DNAとヒストンの複合体)状態の開き具合、が重要であることが分かりつつある。
では、このクロマチンの開き方を調べる手法にはどのようなものがあるだろうか?
今回の記事では、現在主流となりつつあるATAC-seqについて紹介する。
ちなみに、日本人はアタックと呼んでいると思う。海外の人はエィタックと発音すると思う。
-----
ATAC-seq以前に開いたクロマチン領域を調べる方法として使われていたのは、DNase-seq(Genome Research, 2006)やFAIRE-seq(Nature protocols, 2012)である。
DNase-seqはDNAを切断する酵素(DNase)が開いたところだけにアクセスできる、という原理。後述するが、ATACと似ている(ATACが概念的にDNase-seqを真似たのだろうけど。)
FAIRE-seqはホルムアミドでヒストンとDNAを固定するという手法。酵素を使わないので比較的安い。
以下モデル図
しかし、DNase-seqもFAIRE-seqも
- 実験に数日かかる
- いずれも安定した結果を出すにはそれなりの「腕」が必要
という問題があった。
-----
そういうわけで、早く、安定した、技術が求められていた。
これを実現したのがATAC-seq(Assay for transposase-accessible chromatin using sequencing)。
原理的なミソはタグ付きのトランスポゼースである。トランスポゼースはゲノムを切る活性がある酵素。
大事なのは、このトランスポゼースが開いたゲノムにしかアクセスできないこと。ATAC-seqでは、このトランスポゼースにバーコードがついている。
つまり、トランスポゼースを処理してシーケンシングすれば開いたクロマチンのゲノム領域が分かるというわけ。以下の図も参考に。
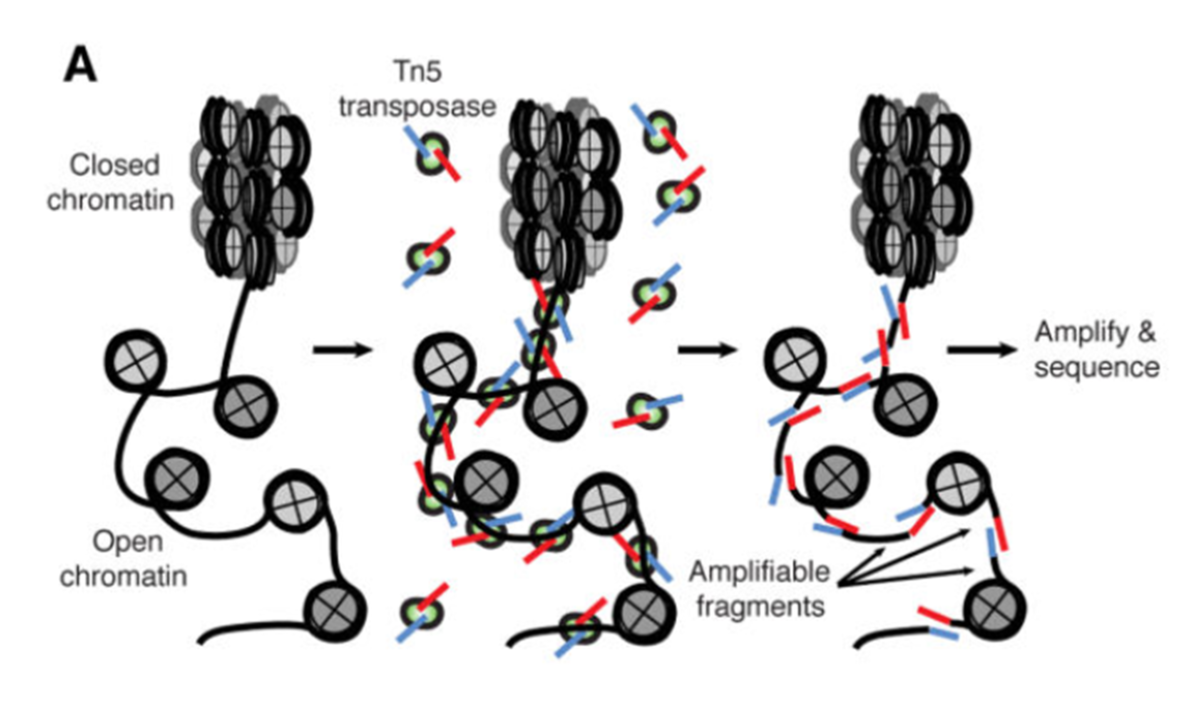
------
これ以降の論文は多すぎて逐一紹介していられない。ちなみにほとんどの論文でシーケンスしていて、qPCRしているものはほとんど見かけない(あるにはあるが)。ATAC-qPCRはあまり安定しないのかも?
また、ATACseqは当然のごとくシングルセルレベルでできるようになっている(Nature, 2015)。
さらに2018年末に10x genomicsがドロップseqと組み合わせたプラットフォームをリリースしていて、数1000細胞のオーダーでも解析できるようになっている。
ついでだと、凍らせたサンプルにも使えるプロトコルも出ている。まあ、細胞はゆっくり凍らせなさい、とかいうことなのだが。
-----
解析パイプラインについてはこちらなどを参照されるといいかも。ほとんどChIP-seqと同じパイプラインが使える。基本的な解析以降はみなさん論文ごとに自由にやっている印象。
-----
参考
- ChIP–seq and beyond: new and improved methodologies to detect and characterize protein–DNA interactions, 2012